Nature Methods|高毅勤/刘思睿团队开发多源实验信息增强的AI模型提升蛋白质复合物结构预测精度
2025年09月22日
蛋白质复合物结构预测是揭示生命机制和推动药物研发的关键技术。尽管AlphaFold等AI模型取得突破,但这些方法在面对大分子复合物、柔性体系及缺乏共进化信号(如抗体-抗原)的蛋白复合体时往往仍面临挑战。现有实验方法如化学交联质谱(XL-MS)、深度突变扫描(DMS)等提供的稀疏约束信息具有重要价值,但现有整合工具存在数据形式单一、约束利用效率低、预测长度限制等不足,影响了预测精度和应用广度。因此,如何发展能够灵活整合多源实验信息的新算法,以实现实验信息增强的AI建模,提升预测精度和效率,成为结构生物学与计算生物学领域共同关注的关键问题。近日,昌平实验室/北京大学高毅勤与昌平实验室刘思睿团队在《Nature Methods》发表发表题为 “Integrating diverse experimental information to assist protein complex structure prediction by GRASP” 的研究论文,提出通用的约束辅助结构预测模型 GRASP(Generalized Restraints Assisted Structure Predictor)。该模型能够灵活整合不同类型和数量的约束信息,从而显著提升蛋白复合物结构预测的精度。

Nature Methods 官网截图
GRASP在AlphaFold-Multimer(AFM)的基础上进行了改进,支持两类通用约束:残基对约束(RPR),如XL-MS或NMR提供的残基间距离信息;界面约束(IR),如共价标记(CL)、化学位移扰动(CSP)或DMS提供的界面残基信息。这些约束被有效整合到相对位置编码、Evoformer和IPA模块中(图1)。此外,团队开发的Combfit电镜密度图建模工具使GRASP能够间接利用电子密度图信息,极大增强了其通用性,使得GRASP天然具备快速高效地同时应用多源实验信息进行整合建模(integrative modeling)的能力。
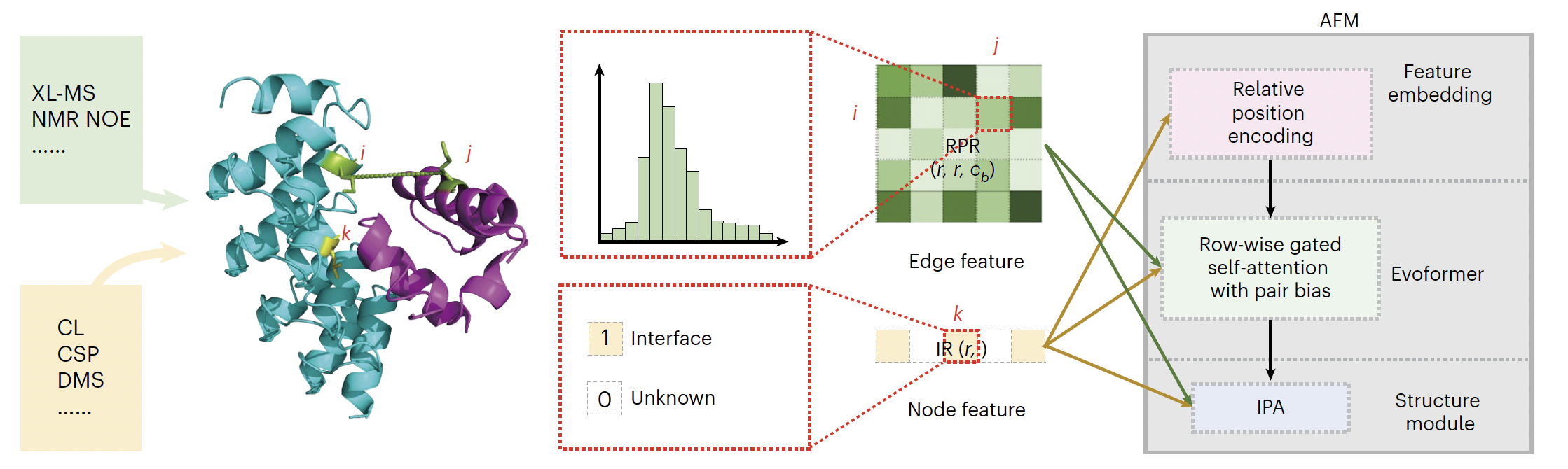
图1. GRASP框架示意图
研究团队在多种模拟与真实数据上验证了GRASP的准确性。在自建的基准集上,GRASP在不同数量和类型的约束下预测表现均优于其他工具,而且能够更好地应用少量约束明显提升预测精度,其建模结构在界面质量和物理合理性上均与实验结构相当。在模拟化学交联数据和单一类型的真实实验数据(如XL、CL和CSP)中,GRASP同样表现出色,并在处理错误约束时展现了良好的鲁棒性。研究团队还展示了GRASP在抗原–抗体复合物上的应用,利用模拟或真实的DMS、XL、CSP约束提升预测精度(图2)。并进一步提出了将GRASP与AF3结合的策略,展现了两者的互补潜力。
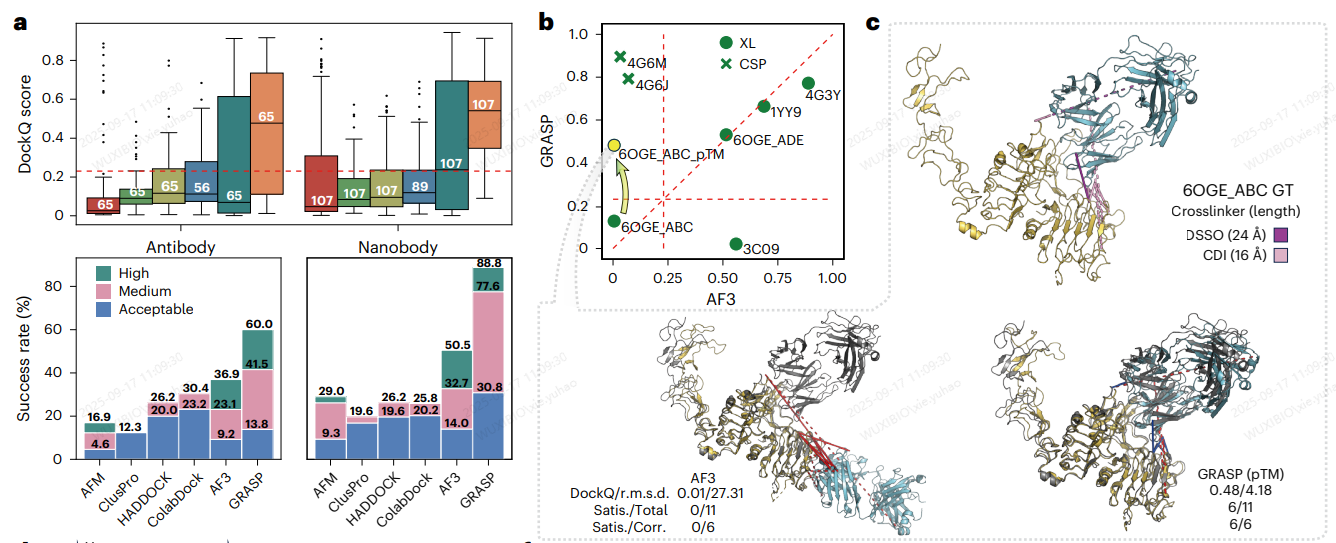
图2. GRASP提高抗原-抗体复合物结构预测精度
研究人员进一步评估了GRASP整合多源实验数据的能力,以及在动态结构或生物原位结构建模方面的潜力。GRASP的模型设计天然适配整合建模任务,与代表性整合建模工具IMP相比,GRASP在整合多样化实验约束(来自交联、突变扫描、冷冻电镜等技术)和提升复合物结构预测精度方面表现更优(图3)。利用原位XL-MS数据,GRASP还可应用于互作组水平的蛋白质对相互作用建模,为原位交联质谱蛋白互作组提供了有力的分析工具,有助于捕捉近天然细胞环境中的瞬时相互作用。GRASP的整合建模与蛋白原位互作结构预测能力展示了它对复杂生物体系与复杂生物问题的适用性。
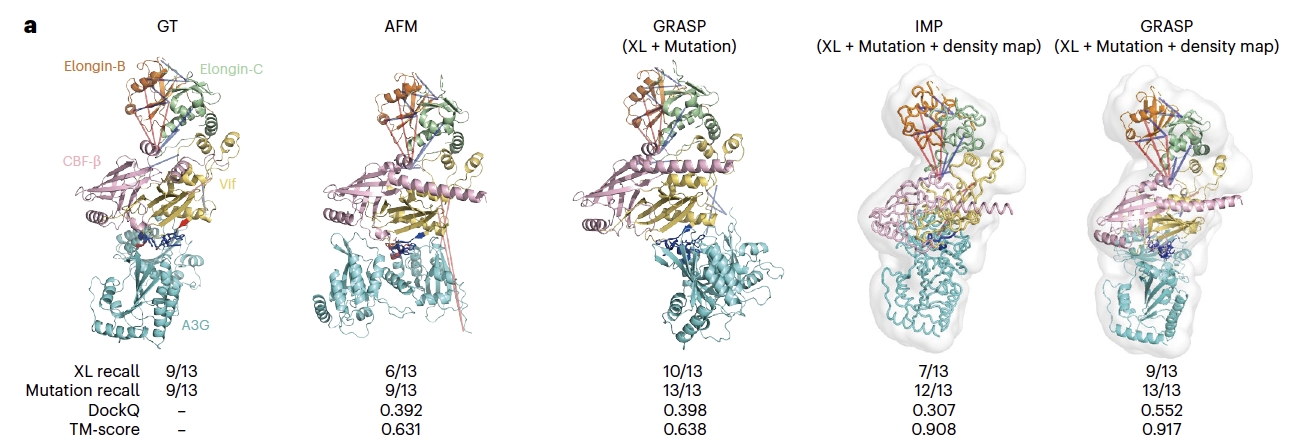
图3. GRASP利用多源实验数据整合建模
在后AF时代,如何充分利用实验信息推动AI与实验技术的协同发展成为重要议题。GRASP通过整合多种实验数据提升了复合物结构预测,特别是对抗原–抗体预测、多源实验信息整合建模和原位PPI重建等问题提供了表现突出的解决方案。该方法有望成为理解与整合分子生物学数据,从而连接实验、计算建模与AI模型的有力工具。GRASP已开源代码和评测数据,并提供可直接运行的Colab Notebook和在线服务器。
北京大学、昌平实验室的高毅勤教授和昌平实验室刘思睿研究员为本文的通讯作者,昌平实验室-北京大学联合培养博士生谢豫豪和北京大学博士生张成伟为本文的共同第一作者。本研究得到了科技部2030重大项目、国家自然科学基金以及新基石科学基金的支持,由昌平实验室、北京大学、华为技术有限公司合作完成。最后,作者们向K. Stahl教授、F. N. Hitawala博士、杨开广研究员、朱祯博士表示感谢。
论文链接: